note d'application sur la métagénomique amplicon 16S/ITS avec microsynth
note d'application sur l'exploration de la composition des communautés microbiennes avec la métagénomique amplicon 16S/ITS

Contenu du document
The Swiss DNA Company
Application Note · Next Generation Sequencing
Introduction
Les communautés microbiennes sont présentes dans presque tous les environnements. Elles sont des moteurs importants des processus biogéochimiques, influencent considérablement la santé humaine et ont un potentiel énorme pour les applications biotechnologiques. Un objectif majeur dans l'analyse d'une communauté microbienne est d'identifier sa composition taxonomique et sa diversité. Le séquençage du gène de l'ARN ribosomal (ARNr) et de ses régions internes de l'espaceur est devenu le standard pour l'identification des micro-organismes tels que les bactéries, les archaea et les champignons. Sa présence dans tous les organismes vivants, la combinaison des régions génétiques conservées et hypervariables et la disponibilité de bases de données de référence référencées en font un marqueur phylogénétique idéal. La combinaison du séquençage de nouvelle génération (NGS) des régions partielles du gène ARNr avec une analyse bioinformatique adéquate est un outil puissant pour explorer la composition des communautés microbiennes.
Choix du système d'amplification
La métagénomique amplicon est basée sur le séquençage NGS du gène ARNr microbien.
Les longueurs de lecture NGS étant limitées, seules des parties du gène de l'ARNr peuvent être amplifiées et séquencées. Pour les procaryotes, l'analyse cible les régions hypervariables (V1-9) du gène ARNr 16S, tandis que pour les champignons, les régions internes de l'espaceur transcrit (ITS) sont utilisées pour le profilage taxonomique. Un système d'amorçage idéal devrait être universel pour couvrir un large éventail de groupes taxonomiques tout en fournissant suffisamment d'informations taxonomiques pour une classification fiable. Sur la base de notre expérience et validation de notre pipeline d'analyse 16S/ITS, nous recommandons les systèmes d'amorçage affichés dans le tableau 1. Notre service n'est pas limité aux gènes marqueurs et systèmes d'amorçage affichés, d'autres gènes marqueurs phylogénétiques (par exemple, cytochrome c oxydase I) et systèmes d'amorçage peuvent également être utilisés. Une étude pilote peut être très utile pour trouver le meilleur système d'amorçage pour votre question de recherche spécifique.
Microsynth Competences and Services
Le profilage des communautés microbiennes est l'une des compétences principales de Microsynth. Basés sur notre vaste expérience, nous fournissons un service de guichet unique allant de la conception expérimentale à l'analyse bioinformatique. Vous pouvez soit externaliser l'ensemble du processus soit seulement certaines étapes. Nous avons validé notre pipeline complet de métagénomique amplicon et les méthodes incluses par séquençage et analyse de deux communautés témoins bien caractérisées. Pour plus d'informations sur notre processus de validation et les possibilités de valider votre propre étude, veuillez nous contacter.
Conception Expérimentale
Le gain et l'impact d'une étude dépendent fortement de sa conception expérimentale. L'utilisation de réplicats, de contrôles et de méthodes d'échantillonnage appropriées ne sont que quelques exemples de points importants à considérer. Profitez de notre expérience ; nos spécialistes NGS vous assisteront dès le début.
Isolation de l'ADN
Les micro-organismes sont hautement divers en ce qui concerne leur physiologie et sont souvent liés à des surfaces comme les particules de sol ou sont organisés en agglomérations stables telles que les biofilms. L'extraction d'ADN à partir de matrices complexes est un défi. Vous pouvez soit réaliser vous-même l'extraction soit externaliser cette étape critique. Microsynth possède une vaste expérience dans l'isolation de l'ADN et de l'ARN à partir de diverses matrices (par exemple, sol ou fèces).
Amplification PCR
L'amplification PCR suit un protocole en deux étapes. Dans une première étape, la séquence spécifique au locus est amplifiée tandis que dans une seconde étape, les adaptateurs de séquençage Illumina et les indices sont ajoutés. Pour les projets avec de très faibles quantités de matériel de départ, nous recommandons notre protocole PCR en trois étapes incluant deux PCR locus-spécifiques successives pour augmenter le rendement des amplicons séquençables.
Séquençage NGS
Le séquençage est effectué sur le système Illumina MiSeq. Le MiSeq permet un profilage à haut débit à faible coût, supportant des longueurs de lecture allant jusqu'à 2x300 bp.
Analyse Bioinformatique
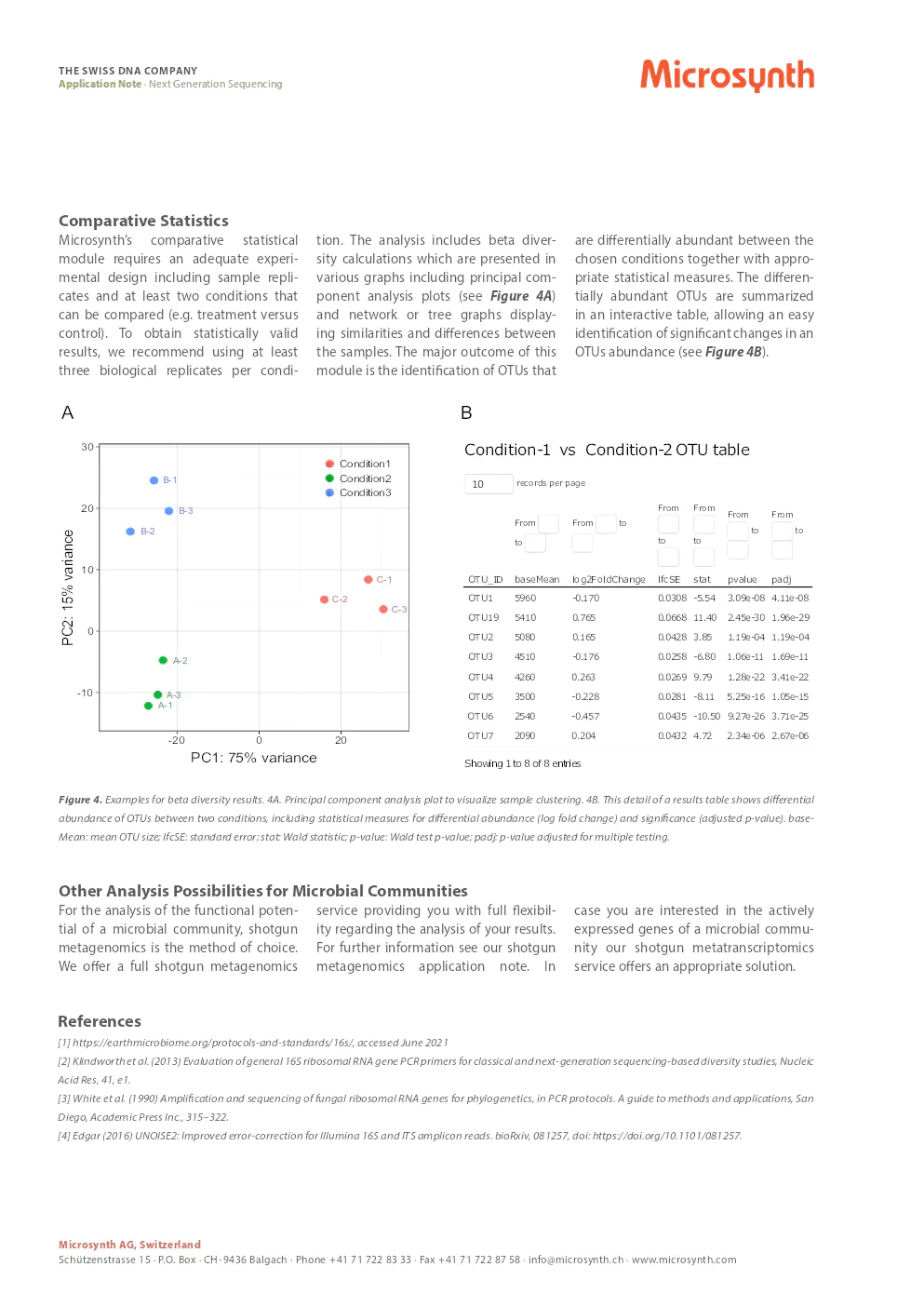
L'analyse de métagénomique amplicon de Microsynth est basée sur des logiciels bioinformatiques à jour et publiés tels que USEARCH pour obtenir des résultats significatifs et fiables pour vos données de séquençage. L'analyse 16S/ITS inclut un filtrage de qualité étendu, un débruitage des unités taxonomiques opérationnelles (OTU), une classification taxonomique basée sur diverses bases de données et une analyse de diversité alpha (c'est-à-dire la diversité OTU au sein d'un échantillon donné). Selon l'objectif de l'étude et la conception expérimentale (conditions, réplicats), Microsynth offre également un module complémentaire pour les statistiques comparatives.
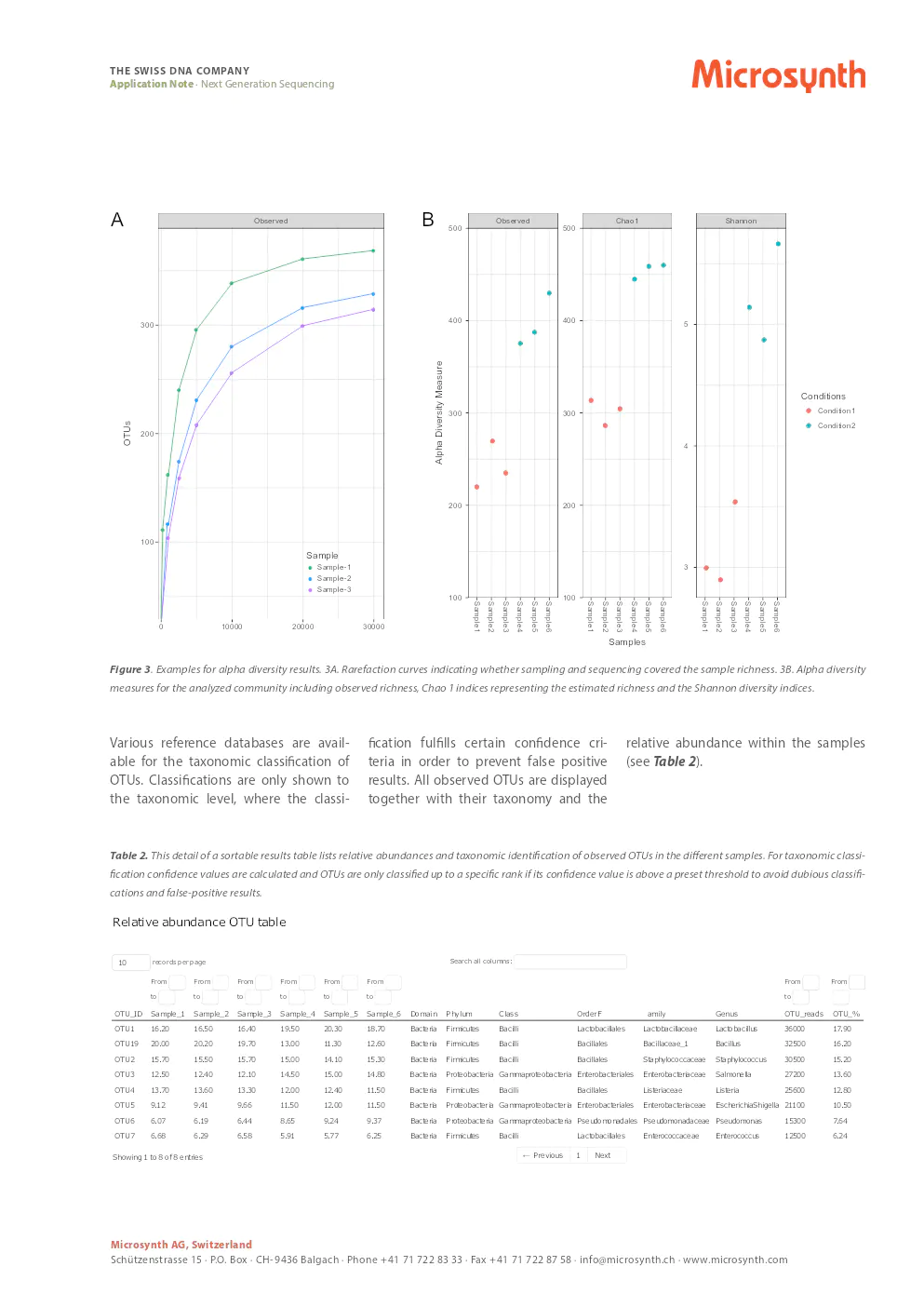
Exemple de Résultats de l'Analyse 16S/ITS
Le pipeline d'analyse de Microsynth vous fournit une caractérisation complète des communautés microbiennes dans vos échantillons. Les mesures de diversité alpha fournissent un aperçu de la richesse en OTU et de la diversité au sein des échantillons.
Microsynth AG, Switzerland
Schützenstrasse 15 · P.O. Box · CH?-?9436 Balgach · Phone +?41 71 722 83?33 · Fax +?41 71 722 87?58 · info@microsynth.ch · www.microsynth.com